CRISPR-AAV系统

CRISPR是一种在细菌中发现的免疫系统,于2013年被成功地应用于真核生物基因组编辑工具中,近年来被广泛应用于基础和临床研究[1],[2],[3]。由于SpCas9敲除小鼠模型的建立和AAV友好型SaCas9和NmCas9[4],[5]的设计出现,AAV和Cas9的组合已成为强大,多功能的动物实验工具[6],[7]。
如有任何问题, 请联系我们免费咨询。
SpCas9
基因敲除
来自MIT的张锋团队培育了SpCas9敲除小鼠,该小鼠可以在全身细胞中表达或Cre依赖性表达SpCas9。研究团队证明利用AAV递送sgRNA和供体序列可以将KRAS基因精确地修饰成肿瘤致癌基因,导致肿瘤在1到3个月内快速生长。SpCas9敲除小鼠现在可以从Jackson Lab处购得。
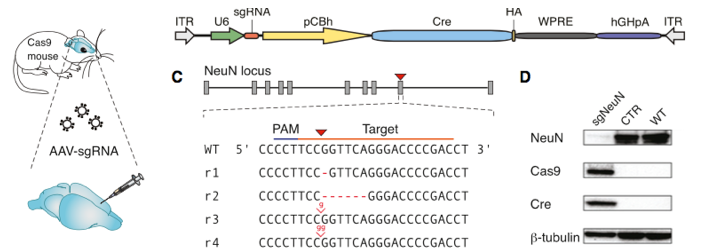
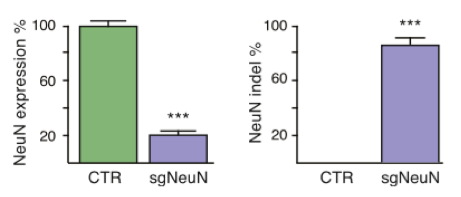
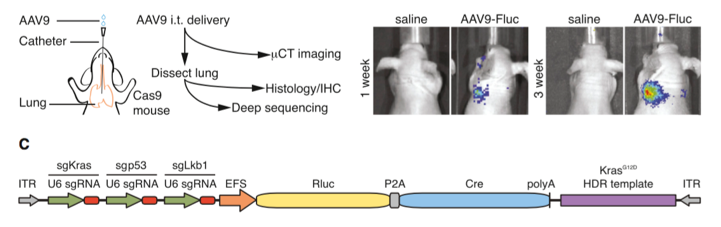
Figures from Ref. 5
基因激活
麻省理工学院的Konermann S.团队发表了他们的成果。 15bp的短sgRNA可以在不切割DNA的情况下将野生型SpCas9锁定在目标基因座上[11]。 因此,在带有两个附加环的sgRNA支架(sgRNA-MS2)结合激活分子MS2-P65-HSF1融合体的情况下,如果sgRNA靶向于转录起始区域,则该基因被激活。该发现提出了一个了克服大基因过度表达而面临AAV载体容量限制的新颖方法。张等人也发现了同样适用于SaCas9的类似修饰[12]。
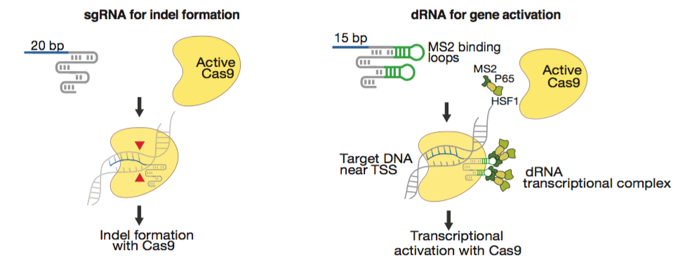
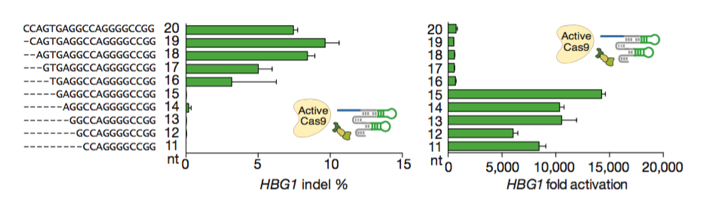
Figures from Ref. 11
派真生物开发了一组sgRNA支架优化的进入质粒,用于创建适合您自己目的的合适构建体。(点击这里)获得等多信息。
SaCas9
AAV的大小限制约为5 Kbp,因此SpCas9的长度(4.3 Kbp)相对较大,不适合灵活操作,比如为不同的靶器官切换合适的启动子。为了更广泛地运用AAV,张等人的小组对SaCas9(3.3 Kbp)进行了改造。与SpCas9相比,SaCas9可以在AAV中以1 Kbp的空间进行特定的表达调控。在一个案例中,AAV用于在肝中特异性表达SaCas9来敲除Pcsk9基因,在这种情况下还观察到40%的靶基因修饰情况。
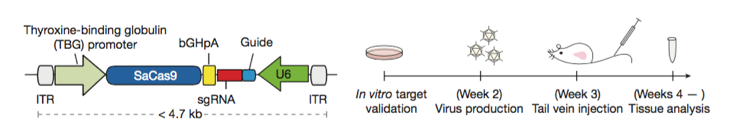
在大多数情况下,使用带有SaCas9的AAV将消除实验对SpCas9小鼠品系的需求,从而节省了在使用疾病小鼠模型进行研究时选取合适小鼠品系杂交的大量时间。
在2015年的最后一天,三个小组分别在Science上发表了他们关于利用AAV将Cas9运用于DMD小鼠治疗的结果[8],[9],[10]。三个小组均采用相同的策略:注射可以表达SaCas9 / SpCas9和sgRNA的AAV,来删除DMD小鼠模型中突变的23号外显子,从而恢复被截短但仍具有功能的肌营养不良蛋白。
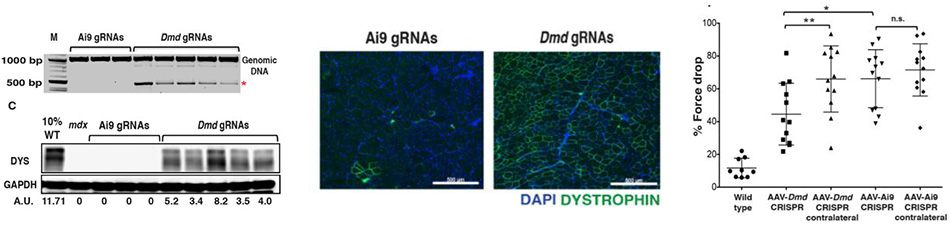
Figures from Ref. 9
对希望在研究中应用这项强大技术的研究人员,派真生物提供了一组优化的入门质粒。
参考文献
[1] Martin Jinek et al. Science 337, 816 (2012); DOI: 10.1126/science.1225829
[2] Le Cong et al. Science 339,819 (2013); DOI: 10.1126/science.1231143
[3] Prashant Mali et al. Science 339,823 (2013); DOI: 10.1126/science.1232033
[4] Randall J. Platt et al. Cell 159, 1–16 (2014); DOI: 10.1016/j.cell.2014.09.014
[5] F. Ann Ran et al. Nature 520, 186 (2015); doi:10.1038/nature14299
[6] Florian Schmidt and Dirk Grimm. Biotechnology J. 10, 258 (2015);[ DOI0.1002/biot.201400529
[7] David B. T. Cox et al. Nature Medicine 21,121 (2015); doi: 10.1038/nm.3793.
[8] C. Long et al., Science 10.1126/science.aad5725 (2015)
[9] C. E. Nelson et al., Science 10.1126/science.aad5143 (2015)
[10] M. Tabebordbar et al., Science 10.1126/science.aad5177 (2015).
[11] Dahlman JE et al., Nat Biotechnol. 2015 Nov;33(11):1159-61.
[12] Nishimasu et al., Cell 162, 1113–1126 (2015)
[2] Le Cong et al. Science 339,819 (2013); DOI: 10.1126/science.1231143
[3] Prashant Mali et al. Science 339,823 (2013); DOI: 10.1126/science.1232033
[4] Randall J. Platt et al. Cell 159, 1–16 (2014); DOI: 10.1016/j.cell.2014.09.014
[5] F. Ann Ran et al. Nature 520, 186 (2015); doi:10.1038/nature14299
[6] Florian Schmidt and Dirk Grimm. Biotechnology J. 10, 258 (2015);[ DOI0.1002/biot.201400529
[7] David B. T. Cox et al. Nature Medicine 21,121 (2015); doi: 10.1038/nm.3793.
[8] C. Long et al., Science 10.1126/science.aad5725 (2015)
[9] C. E. Nelson et al., Science 10.1126/science.aad5143 (2015)
[10] M. Tabebordbar et al., Science 10.1126/science.aad5177 (2015).
[11] Dahlman JE et al., Nat Biotechnol. 2015 Nov;33(11):1159-61.
[12] Nishimasu et al., Cell 162, 1113–1126 (2015)

