
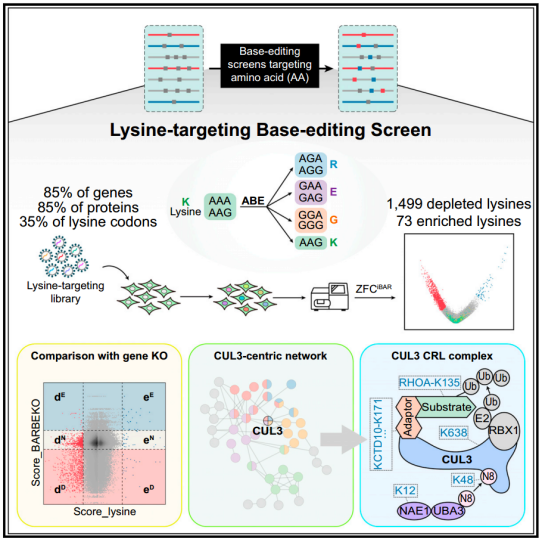
2023年11月22日,北京大学魏文胜、周卓共同通讯在Molecular Cell 在线发表题为“Unbiased interrogation of functional lysine residues in human proteome”的研究论文,该研究建立了一种CRISPR策略,用于在基因组尺度上无偏倚地探测功能氨基酸残基。
通过结合腺嘌呤碱基编辑器和条形码sgRNAs,研究人员在611.267个赖氨酸密码子中筛选出215.689个(35%),涉及85%的蛋白质编码基因。鉴定出1572个赖氨酸密码子,它们的突变扰乱了人类细胞的适应性,其中许多与癌症有关。然后将这些密码子镜像到基因敲除筛选数据中,以提供对赖氨酸残基在细胞适应性中的作用的功能见解。挖掘这些数据,作者发现了一个以CUL3为中心的调节网络,其中CUL3 CRL复合体蛋白的赖氨酸残基通过指定蛋白质-蛋白质相互作用来控制细胞适应性。该研究提供了一种查询遗传元素的一般策略,并提供了对人类蛋白质组的功能见解。

氨基酸(AA)残基是蛋白质结构和活性的基础。氨基酸的遗传取代与生理变化或疾病有着广泛的联系,在所有已知的致病性突变中所占的比例很大。因此,在某些生理或病理背景下系统地询问AA残基是有价值的。目前,这主要是通过低吞吐量的方式完成的。
最近开发的CRISPR-Cas介导的碱基编辑技术(BE)在DNA水平上实现了单碱基替换。碱基编辑器包括腺嘌呤碱基编辑器(ABEs)和胞嘧啶碱基编辑器(CBEs),它们分别将A、T碱基对转化为G、C碱基对和C、G碱基对转化为T、A碱基对。由于BEs能够在各种生物体中有效地安装点突变,它们适用于高分辨率的高通量遗传筛选。最近,BEs已被利用在汇集筛选中来调查哺乳动物细胞中的单核苷酸变异(SNVs)或绘制特定蛋白质的功能残基,识别出许多参与不同途径的功能变异。然而,这些研究只包括特定的基因或有限的SNVs。
机理模式图(图源自Molecular Cell )
由于真核蛋白的中位长度为361 AA,因此用于靶向的遗传密码子总数是基因数量的数百倍,这给全基因组文库的处理带来了挑战。因此,研究人员专注于一个特定的AA残基。赖氨酸残基是这种研究的有吸引力的目标,因为它们是多种翻译后修饰(PTMs)的主要受体,如泛素化和乙酰化,这对调节蛋白质的功能和稳定性至关重要。
该研究提出了一种全基因组筛选功能性AA残基的策略。与传统的CRISPR KO筛选不同,该策略可以精确定位蛋白质的功能残基,为机制研究提供直接指示。此外,由于AA取代可以降低或增加蛋白质丰度,失活或激活蛋白质功能,此策略允许在单个筛选中同时进行功能获得和功能丧失的扰动。此外,这一方法可以检测赋予蛋白质独特活性的突变。这些突变可能与致病过程有关,而其对应的基因无法通过CRISPR KO或CRISPRa筛选鉴定。因此,这种方法为获得人类蛋白质组的功能见解提供了前所未有的机会。
北京大学魏文胜课题组博士后宝颖、博士研究生潘倩和已毕业的许萍博士和刘志恒博士为论文的共同第一作者,魏文胜教授和副研究员周卓(现为中国医学科学院系统医学研究院/苏州系统医学研究所研究员)为论文的共同通讯作者。该研究获得了国家重点研发计划、国家自然科学基金、北京市科委生物医学前沿创新推进项目、中国医学科学院医学与健康科技创新工程、北大-清华生命科学联合中心、昌平实验室、重大疾病共性机制研究全国重点实验室等的支持。
资料来源
1.公众号iNature
https://mp.weixin.qq.com/s/v3QahJFBeqiLn9QHt3YgzQ
关于派真
作为一家专注于AAV 技术十余年,深耕基因治疗领域的CRO&CDMO,派真生物可提供从载体设计、构建到 AAV、慢病毒和 mRNA 服务的一站式解决方案。凭借深厚的技术实力、卓越的运营管理和高标准的服务交付,我们为全球客户提供一站式CMC解决方案,包括从早期概念验证、成药性评估到IIT、IND及BLA的各个阶段。
凭借我们独立知识产权的π-alphaTM 293 细胞AAV高产技术平台,我们能将AAV产量提高多至10倍,每批次产量可达1×10¹⁷vg,以满足多样化的商业化和临床项目需求。此外,我们定制化的mRNA和脂质纳米颗粒(LNP)产品及服务覆盖药物和疫苗开发的各个阶段,从研发到符合GMP的生产,提供端到端的一站式解决方案。

