主要研究结果
2023年6月27日,中国科学院遗传与发育生物学研究所高彩霞研究组在 Cell 期刊发表了题为:Discovery of deaminase functions by structure-based protein clustering 的研究论文。
该研究创新性地运用AI辅助的大规模蛋白结构预测,建立起全新的基于三级结构的高通量蛋白聚类方法,实现了脱氨酶功能结构的深入挖掘,鉴定到完全区别于已知脱氨工具酶的全新底盘元件,并成功开发了一系列具有我国自主知识产权的新型碱基编辑工具。派真生物很荣幸能为该研究提供AAV病毒包装服务,助力新型碱基编辑工具开发。

研究团队首先通过蛋白质结构预测模型AlphaFold2对具有代表性的脱氨功能序列进行批量三维结构预测,随后创新性地开展了基于三维结构的蛋白质多重比对与聚类,成功将潜在的脱氨酶划分为20个不同的分支。除已报道的APOBEC/AID胞嘧啶脱氨酶外,研究人员又检测到了5个结构、序列全新的具有活性的胞嘧啶脱氨酶分支。在这些分支中,研究团队对具有类DddA (Double-stranded DNA deaminase toxin A-like)脱氨结构域的蛋白进行进一步结构聚类和功能验证,发现除以前推测的具有双链DNA脱氨活性的蛋白外,该分支还包含了大量只具有单链DNA脱氨活性的蛋白,该结果颠覆了之前对该类蛋白功能的认知。
以上研究表明,当蛋白集合的序列同源性较低且功能多样时,相比于传统的基于氨基酸一级序列的聚类方法,通过AI辅助的蛋白质结构聚类能够得到更准确的结果。因此,该方法为蛋白质功能分析和挖掘提供了一个高效、可靠的新策略。
基于上述进一步聚类的结果,全新鉴定到45个单链胞嘧啶脱氨酶(Sdd)和13个双链胞嘧啶脱氨酶(Ddd)。这些脱氨酶是目前唯一全部来自于原核生物(细菌)的脱氨酶,而现有APOBEC/AID脱氨酶家族成员都来自于真核生物(主要包括人、哺乳动物或鱼类)。
研究团队基于这些脱氨酶开发了一系列新型碱基编辑系统,并在动、植物细胞中进行了测试。结果表明,新开发的基于Ddd1和Ddd9脱氨酶的双链碱基编辑系统克服了常规编辑器对GC序列编辑效率明显降低的缺陷;基于Sdd7和Sdd3的单链碱基编辑系统展现出了非常高的编辑活性,在GC序列同样具有可观的碱基编辑能力;基于Sdd6的单链碱基编辑系统则展现出了极高的特异性,几乎检测不到脱靶事件。
研究团队进一步通过蛋白理性设计和功能验证,开发了新型的可被单个腺相关病毒(AAV)包被的Sdd6-CBE碱基编辑器,在小鼠细胞系中成功获得高达43.1%的编辑效率, 解决了常规碱基编辑器过大而无法被腺病毒颗包被递送的难题。此外,针对大豆中长期存在碱基编辑效率低下的问题,研究团队新开发了Sdd7-CBE系统,在154株大豆阳性苗中获得了34株稳定编辑的植株,编辑效率高达22.1%。这些脱氨酶是目前唯一全部来自于原核生物(细菌)的脱氨酶,而现有APOBEC/AID脱氨酶家族成员都来自于真核生物(主要包括人、哺乳动物或鱼类)。
该研究突破了现有脱氨酶的应用瓶颈,展现出新型碱基编辑系统在医学和农业方面广泛的应用前景。
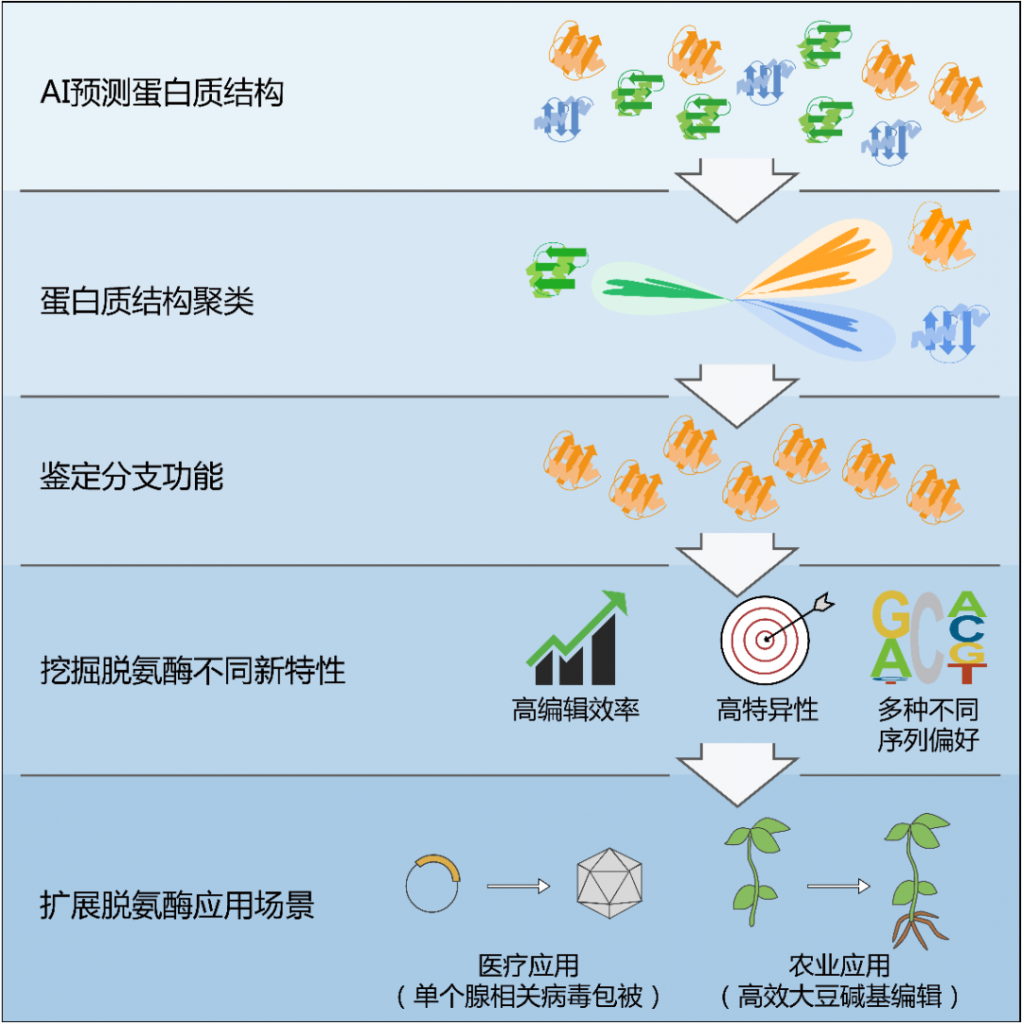
基于AI辅助的蛋白结构聚类挖掘脱氨酶并开发具有新特性的碱基编辑系统
研究小结
综上所述,该项工作为蛋白功能分析、新功能元件挖掘提供了全新策略。新研发的碱基编辑系统是具有我国自主知识产权的精准基因编辑技术,有望打破碱基编辑底层专利垄断,将帮助我国在未来的生物技术产业竞争中处于有利地位。
直播回看
为搭建派真及行业技术专家与各领域科研人员沟通互动的桥梁,让大家学习交流前沿的科研进展与研究思路,派真生物特推出Π学院【与SCI一作对话】系列直播,邀请一作与大家分享他们在基因治疗等领域的最新重要研究成果,希望能够帮大家增进对基因治疗前沿研究的理解,同时找到自己的研究思路。
2023年9月12日,派真有幸邀请到Cell一作黄佳颖老师就该篇论文分享,内含满满干货。错过直播的朋友们也不用着急,小派有好好保存当日记录,后台回复关键词【回看】,即可回顾直播详情~

