研究背景
先导编辑是一种革命性的基因编辑技术,能够在不“切割”两条DNA链的情况下,实现精确的碱基替换、小片段的插入和删除。先导编辑系统是一种由两种成分组成的复合物。其中一个组件是先导编辑器(Prime editor, PE),它结合了Cas9切口酶(第一个CRISPR-Cas基因编辑技术用到的核酸酶)和逆转录酶(RT,一种将RNA逆转录成DNA的酶)。另外一个组成成分是引导RNA(prime editing guide RNA, pegRNA),这是一种经过修饰的RNA,可以识别DNA中的目标序列,在基因编辑过程中起向导RNA和DNA模板的作用。在这个综合体中,pegRNA就像“导航”,而PE则像“文字处理机”,在pegRNA的引导下,PE可以准确地替换基因组信息。目前该技术已被广泛应用于基因功能研究、疾病模型构建和基因治疗中,但该技术的体内基因编辑效率波动大,因此急需进行优化。先导编辑工具的具体优化策略包括:PE相关酶的改良、pegRNA优化和递送策略的创新等。
由于免疫原性低、宿主范围广泛且易于生产,腺相关病毒(AAV)被认为是将PE系统递送至靶器官/组织进行体内基因治疗的最合适载体之一。然而,现有的PE体积较大,超出了AAV载体的容量,这限制了其通过AAV在体内的高效递送。为了克服这个限制,研究人员探索了多种策略,包括断裂内含子(split inteins)、MS2、SunTag和直接分离式PE(sPE)系统等,但这些策略要么过程复杂,要么可能会影响编辑效率。
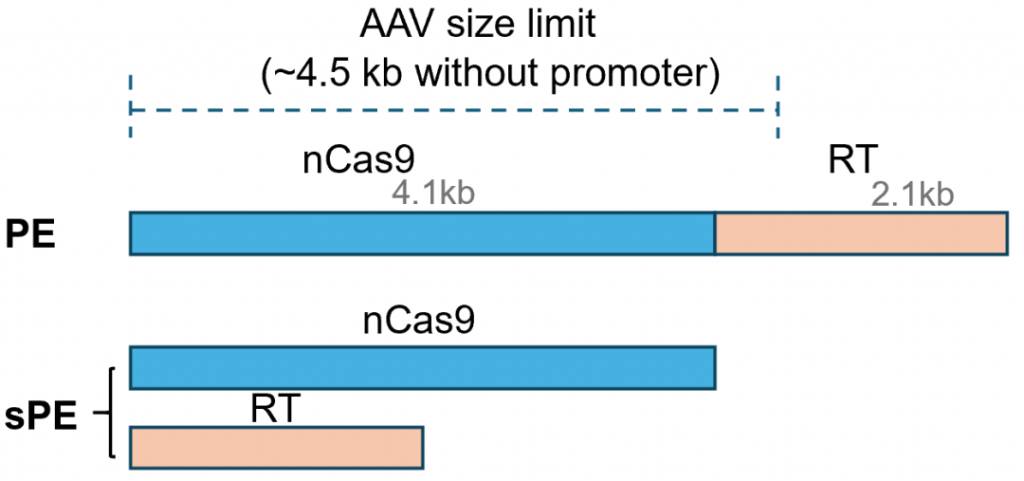
图1 PE和sPE结构示意图
近日,中国科学院广州生物医药与健康研究院赖良学团队、王可品团队和暨南大学陈孟龙团队联合在Genome Biology杂志(IF 12.3)发表研究论文,利用卷曲-螺旋二聚体(CC)肽通过静电和疏水相互作用可形成紧密且特异性的二聚体配对这一特性,开发了一种结构架构相对简单灵活的PE系统(CC-PE),显著提高了基因编辑效率,特别适用于治疗应用。

>>>
研究亮点
开发了一个基于卷曲螺旋结构的先导编辑器(CC-PE),与融合的先导编辑器和现有的分离式先导编辑器相比,CC-PE更简单、灵活,体外和体内的基因编辑效率更高,特别适用于治疗应用。
>>>
研究思路
CC-PE的设计
体外基因编辑性能评价(编辑效率、indel副产物水平、脱靶率等)
体内基因编辑性能评价(编辑效率、靶基因的表达水平等)
>>>
研究结果
为了构建CC-PE,作者首先将短肽P3或N5融合到nCas9的C末端,并将P4或N6连接到RT的N末端,在HEK293T细胞中的28个基因位点上比较了CC-PE与未分离PE以及四种先前报道的分离策略的编辑效率,发现CC-PE在大多数基因组位点上的精确编辑效率显著提升,而indel副产物比例并没有提高。接下来,作者又在A549、HCT116和U2OS三种人类细胞系验证了CC-PE的适用性,结果表明CC-PE的编辑效率通常超过了直接分离PE系统,与融合PE系统相当甚至更优。
由于SpCas9具有NGG PAM偏好,因此使用传统的先导编辑器能够靶向的基因组区域会受到限制,而将改进的Cas9变体(如具有NG PAM的SpCas9-NG和具有NRN PAM的SpCas9-SpRY)整合到先导编辑器中,能够扩展基因组的编辑范围。因此,作者继续设计了两种具有更灵活PAM的CC-PE:NG-CC-PE和SpRY-CC-PE,在9个NG PAM目标位点上检测了编辑效率。结果表明,将先前鉴定的SpCas9变体整合到CC-PE编辑器中可以放宽传统的PAM偏好,从而扩展了CC-PE的潜在应用。
作者接下来验证了CC-PE能否通过双AAV灵活递送并在体内实现精准编辑。为了最大化体内先导编辑效率,作者对pegRNAs的3’末端进行了evopreQ1修饰(EpegRNA),以增强pegRNA的稳定性,提高先导编辑效率。以小鼠Pcsk9为靶标基因,设计pegRNA以及相应的切割sgRNA,通过在Pcsk9基因的第一外显子插入终止密码子(TGA)或T以敲除Pcsk9.结果显示sPE-P3P4注射组小鼠的总胆固醇和LDL胆固醇水平显著降低,且小鼠的丙氨酸氨基转移酶(ALT)和天门冬氨酸转移酶(AST)水平没有升高,表明没有明显的肝细胞损伤。总的来说,这些数据强有力地证明CC-PE可以被AAV有效递送用于体内精准基因编辑。

图2 体内对Pcsk9进行先导编辑及其对血浆胆固醇水平的影响
>>>
结论
0
开发了一个灵活版本的PE,通过卷曲螺旋二聚体化肽将nCas9和RT耦合起来,这些肽只包含28个氨基酸,几乎不会增加载体大小。与之前报道的分离策略(包括断裂内含子、MS2和SunTag)相比,CC-PE的构建过程要简单得多。
0
CC-PE具有更高的体外和体内基因编辑效率,特别适用于治疗应用。
派真助力:AAV病毒包装和生产
参考资料:
1. https://doi.org/10.1089/hum.2020.231
2. https://doi.org/10.1186/s13059-024-03257-z
3. https://doi.org/10.1016/j.scib.2023.11.015
